2020年のノーベル化学賞は、「The development of a method for genome editing」の業績に対して、Emmanuelle CharpentierとJennifer A. Doudnaに与えられた。「CRISPR-Cas9を用いたゲノム編集技術」は生命の設計図であるゲノム情報の改変を可能とし、生命科学の基礎研究から動植物の品種改良や遺伝子治療といった応用にいたるさまざまな分野において利用されている。CRISPR-Cas9は原核生物の免疫システムに関する基礎研究の中で発見されたRNA依存性DNA切断酵素であり、基礎研究が技術革新につながった好例といえる。さらに、世界中の研究者が、微生物学、生命情報科学、分子生物学、生化学、構造生物学、細胞生物学などさまざまな手法を駆使してCRISPRの機能の解明に挑み徐々に謎が解けていくプロセスはまさにサイエンスの醍醐味である。ここではCRISPRの発見からゲノム編集への応用までの研究の歴史を紹介したい。
CRISPR-Cas獲得免疫システム
まず、CRISPR-Cas獲得免疫システムの概要を説明する (図1)。多くの原核生物 (真正細菌および古細菌) のゲノムにはCRISPR (clustered regularly interspaced short palindromic repeats) とよばれる特徴的な繰り返し配列が存在する (最近の研究から、メガファージもCRISPRをもつことが報告されている1)。CRISPRはリピートとスペーサーとよばれる配列の繰り返しからなる。リピートは長さと塩基配列が一定である一方、スペーサーは長さが一定のランダムな塩基配列 (外来核酸に由来する任意の配列) である。1987年に最初のCRISPR (当時はCRISPRとはよばれていなかったが) として発見された大腸菌由来CRISPRは、29塩基のパリンドローム配列をもつリピートと32塩基のランダム配列をもつスペーサーからなる2。CRISPRの近傍には、Cas1やCas2などのCas (CRISPR-associated) タンパク質をコードするcas オペロンが存在する。原核生物に感染した外来核酸 (プラスミドやバクテリオファージ) はCas1-Cas2複合体によって断片化され、あらたなスペーサーとしてCRISPRに取り込まれる。CRISPRから転写されたcrRNA (CRISPR RNA) は特定のCasタンパク質と複合体を形成する。スペーサーに由来するcrRNAのガイド配列は過去に感染した外来核酸の配列と一致するため、再感染した外来核酸と塩基対を形成することができる。したがって、Cas-crRNA複合体は再感染した外来核酸を特異的に認識することができる。なお、後述するが、Cas9など一部のCasタンパク質は2種類のRNA (crRNAとtracrRNA) と結合する。現在、CRISPR-Cas獲得免疫システムは6つのタイプ (I~VI型) に分類されており、Cas9はII型CRISPR-Casシステムに関与するRNA依存性DNA切断酵素である3。
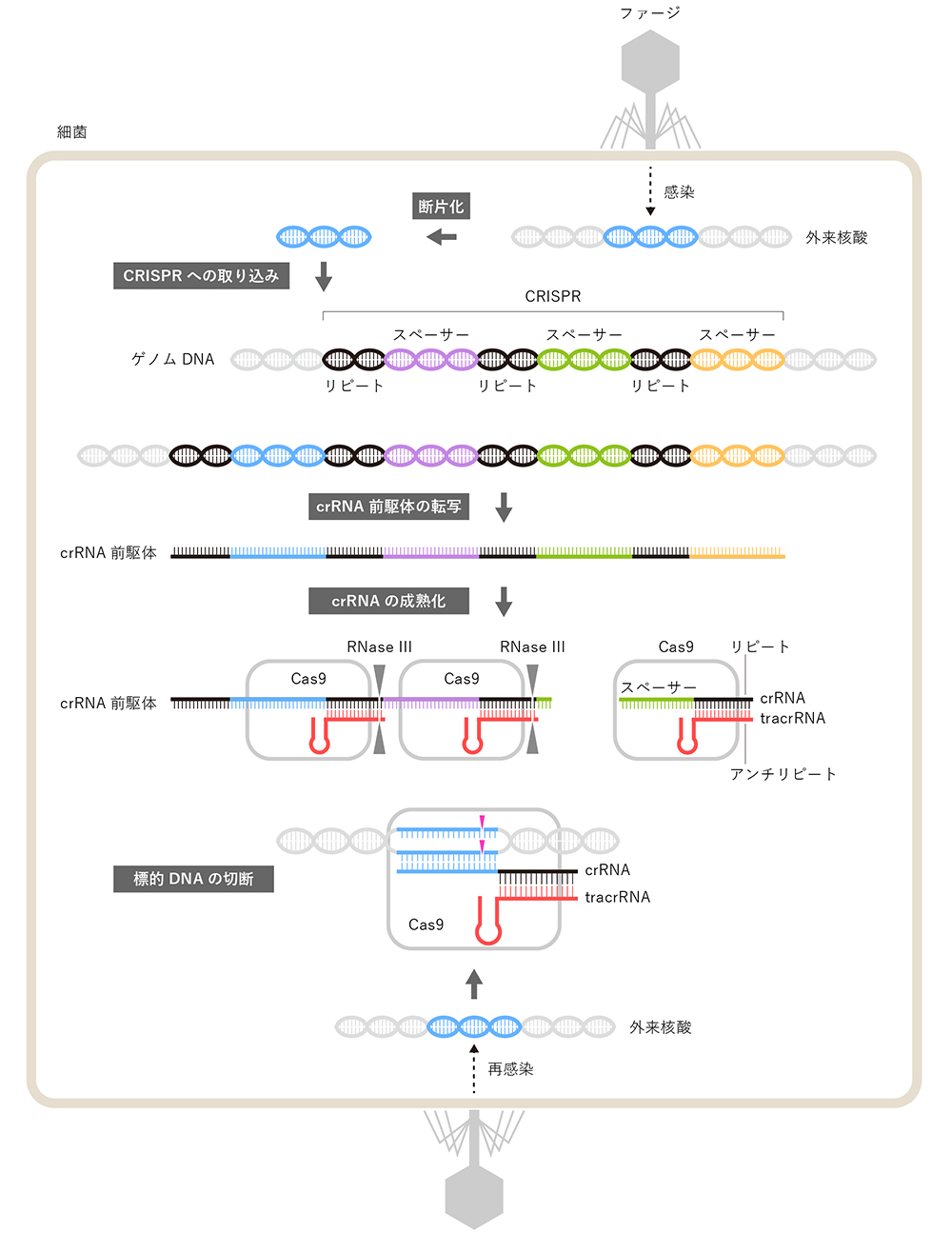
図1 II型CRISPR-Casシステム
CRISPRの発見
CRISPRは30年以上前に偶然発見された。1987年、石野 (現・九州大学教授) らは、大腸菌のiap遺伝子 (isozyme conversion of alkaline phosphatase; アミノペプチダーゼ) の塩基配列解析の過程において、iap遺伝子の近傍に存在する奇妙な繰り返し領域 (CRISPR) を発見した2。しかし、その生物学的な役割は不明であり、論文は、“So far, no sequence homologous to these has been found elsewhere in procaryotes, and the biological significance of these sequences is not known.”という一文で締めくくられている。CRISPRの発見に関しては石野先生ご自身が書かれた記事に詳しい。
CRISPRの機能
2000年代になると多くの生物種のゲノム配列が解読され、配列解析が可能となった。Francisco MojicaやRuud Jansenらは、同様の繰り返し配列が多くの原核生物において保存されていることを発見し、CRISPRと命名した4。2005年、Mojicaらは、67種の原核生物に由来する約4500個のスペーサーの配列解析を行い、スペーサーは外来核酸と配列同一性をもつことを報告した5。さらに、古細菌Sulfolobus solfataricus はSIRVウイルスに感染しないという先行研究の結果、および、S. solfataricus はSIRVウイルスの一部と一致する配列をCRISPRのスペーサーとしてもつという観察事実にもとづき、CRISPRが獲得免疫に関与している可能性を提唱した。また、真核生物のRNA干渉との比較から、CRISPRはRNAとして転写されて機能する可能性も示唆された。しかし、CRISPRが獲得免疫に関与している実験的な証拠はなかった。
2007年、ダニスコ社のRodolphe Barrangou、Philippe Horvathらは、乳酸菌Streptococcus thermophilus に感染したファージのDNAがスペーサーとしてCRISPRに取り込まれることを報告した6。さらに、分子生物学的解析を行い、S. thermophilus はスペーサーと一致するDNA配列をもつファージに抵抗性を示すこと、および、この抗ウイルス活性にはcas9 遺伝子 (当時はcas5 とよばれていた) が必要であることを示した。この研究によって、CRISPRの生理的な役割、すなわち獲得免疫としての機能が実験的に明らかになった。
2008年、John van der Oostのグループは、生化学的解析により、大腸菌のCRISPRはcrRNAとして転写されること、および、crRNAは5種類のCasタンパク質と巨大 (405 kDa) な複合体 (CRISPR-associated complex for antiviral defense (Cascade) と命名) を形成することを明らかにした7。分子生物学的解析の結果、CascadeおよびCas3ヌクレアーゼ/ヘリカーゼが抗ウイルス活性に必要であることが示唆された。2012年、同グループは生化学的解析により、Cascadeが標的DNAに結合すると、Cas3がリクルートされDNAの巻き戻しと分解が引き起こされることを報告した8。2014年には複数のグループによってCascadeの立体構造が決定され、I型CRISPR-Casシステムの作動機構の理解が進んだ9-11。crRNAのリピート領域 (パリンドローム配列をもつ) は実際にヘアピン構造をとりCas6サブユニットによって認識されていた一方、ガイドとしてはたらくスペーサー領域は標的DNAと対合できるように溶媒側に露出していた。これらの結果から、CRISPRの命名の由来である「palindromic repeats」の役割が説明された。ただし、後述するように、Cascade-Cas3が関与するI型CRISPR-Casシステムと異なり、Cas9が関与するII型CRISPR-Casシステムにおいては、crRNAのリピート領域はパリンドローム配列をもたずtracrRNAと対合する。したがって、II型CRISPR-Casシステムの繰り返し配列にもとづき命名されていたら、「CRISPR」という名称ではなかっただろう (多くの原核生物はI型CRISPR-Casシステムをもち、II型CRISPR-Casシステムはレアであるので、その可能性はほとんどないが)。
II型CRISPR-CasシステムにおいてCas9はどのようなメカニズムで獲得免疫に関与しているのだろうか?2010年、Sylvain Moineauのグループは、S. thermophilus に感染した外来核酸 (2本鎖DNA) はスペーサーと相補的な領域で切断されることを報告した12。標的DNAの切断にはcas9 遺伝子が必須であることも明らかになった。Cas9は既知のDNAエンドヌクレアーゼ (RuvCとHNH) と相同なモチーフをもつことがわかっていた。これらの結果から、I型CRISPR-Casシステムに関与するCascade-Cas3はシュレッダーのようにはたらき標的DNAをバラバラに分解するのに対し、II型CRISPR-Casシステムに関与するCas9-crRNA複合体 (実際はCas9-crRNA-tracrRNA複合体) はハサミのようにはたらき標的DNAを「きれいに」切断することが示唆された。
tracrRNAの発見
2011年、Charpentier、Jörg Vogelの共同チームは、II型CRISPR-Casシステムが抗ウイルス活性を発揮するには、Cas9とcrRNAに加えて、tracrRNA (trans -activating crRNA) が必要であることを報告した13。ちなみに、crRNAはシーアールRNA、tracrRNAはトレーサーRNAと発音されることが多い。Charpentierらは、化膿レンサ球菌Streptococcus pyogenes においてCRISPRの近傍から未知のRNA (tracrRNA) が大量に転写されていることを発見した。シーケンス解析および分子生物学的解析の結果、crRNA前駆体とtracrRNAはコプロセシングされることが示唆された。この結果と一致して、tracrRNAは、crRNAのリピート領域と相補的な領域 (アンチリピートと命名された) をもっていたことから、crRNAとtracrRNAは対合することが示唆された。Charpentierらは、内在性のRNase III (2本鎖RNA切断酵素) がcrRNA:tracrRNAのコプロセシングに関与するのではないかという仮説を立て、分子生物学的解析および生化学的解析を行い、Cas9、tracrRNA、および、RNase IIIがcrRNAの成熟化に必要であることを明らかにした。これらの結果から、crRNA前駆体はtracrRNAと対合した状態でCas9に結合し、RNase IIIによって切断され、crRNAが産生されることが示唆された。2011年の時点では、tracrRNAは主にcrRNAの成熟化に関与すると考えられており、論文には“Csn1 may also be involved in the silencing of invading sequences.”と記載されている (当時Cas9はCsn1とよばれていた)。研究が進んでいたI型CRISPR-CasシステムではcrRNAのみがガイドRNAとして関与するため、tracrRNAの発見は予想外でありII型CRISPR-Casシステムの理解に大きく貢献した。
「The Heroes of CRISPR」14によると、CharpentierらはII型CRISPR-Casシステムの「ミッシングピース」の同定を意図していたわけではなく、S. pyogenes の病原性を制御するRNAの同定を目指し超並列シーケンス解析を行う過程においてtracrRNAを発見したらしい。「The Heroes of CRISPR」は、内容に偏りがあるということで物議を醸したが、CRISPRの発見から応用にいたるまでの舞台裏や関連研究者のキャリアなど貴重な情報が書かれているので、ぜひ一読をおすすめする。
CRISPR-Cas9の生化学的解析
以上の研究から、II型CRISPR-CasシステムではCas9、crRNA、および、tracrRNAが標的DNAの切断に関与することが示唆されていたが、直接的な証拠はなかった。Doudnaの著書「A Crack in Creation」(と「Heroes of CRISPR」) によると、2011年3月、プエルトリコで開催された国際学会においてCharpentierとDoudnaは出会い共同研究を開始した。2012年6月、共同チームは、精製したCas9タンパク質、crRNA、tracrRNA、標的DNAを用いた生化学実験を行い、Cas9-crRNA-tracrRNA複合体が標的DNAを切断することを報告した15。この結果、tracrRNAはcrRNAの成熟化だけでなく、標的DNAの切断にも関与することが明らかになった。今となってはtracrRNAがDNA切断にも関与することに気づくのは難しくないようにも思えるが、当時はそれほど簡単ではなかったようだ。筆頭著者のMartin Jinek (筆者と同い年で、Cas9の構造解析を通じて親友になった) に聞いてみたところ、最初はCas9-crRNA複合体を用いて標的DNAの切断を調べていたとのことだった。
さらに、JinekらはcrRNAとtracrRNAをテトラループで連結したキメラRNA (sgRNA (single-guide RNA) と命名された) がin vitro においてcrRNA:tracrRNAと同様にはたらくことを発見した。ただ、この論文において提唱されたsgRNAは、tracrRNAの3′側の2つのステムループ (tracrRNA tail) が削除された短縮型であり、in vitro におけるCas9との結合および標的DNAの切断には十分だが、真核細胞におけるゲノム編集には活性が不十分であることがわかっている16。Jinekらはin vitro においてガイド配列を変更することにより標的DNAを変更できることも示した。これらの生化学的解析の結果、Cas9-sgRNA複合体は「プログラム可能な」DNA切断酵素としてはたらき、ゲノム編集に利用できる可能性が示唆された。論文は、“We propose an alternative methodology based on RNA-programmed Cas9 that could offer considerable potential for gene-targeting and genome-editing applications.”という一文で締めくくられている。
2012年9月、Virginijus Siksnysのグループは、S. thermophilus に由来するCas9およびCRISPRアレイ (crRNAとtracrRNA) を大腸菌において共発現させ、精製した複合体が標的DNAを切断することを明らかにし、Cas9-crRNA複合体が標的DNAを切断すると報告した17。実際にはこの複合体は (CRISPRアレイを共発現させているため) tracrRNAをふくむCas9-crRNA-tracrRNA複合体であったが、tracrRNAの存在は調べられていなかった。論文のタイトルも「Cas9–crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria」だった。ただ、「Heroes of CRISPR」によると、Siksnysらも、精製したCas9タンパク質、crRNA、tracrRNAを用いた生化学実験を行い、tracrRNAが標的DNAの切断に必須であることを見出していたが、 (理由は不明だが) tracrRNAに関するデータは最終的に論文に含めなかったらしい (2012年3月に出願された特許には記載されている)。
CRISPR-Cas9を利用したゲノム編集
2013年1月、Feng Zhangのグループは、CRISPR-Cas9を用いたゲノム編集技術を報告した18。Zhangらは、標的遺伝子と相補的なガイド配列をもつガイドRNA (crRNA:tracrRNA) およびS. pyogenes 由来Cas9をヒト培養細胞に共発現させ、標的遺伝子の塩基配列を改変できることを報告した。さらに、(1) ガイド配列の異なる2種類のガイドRNAを用いることにより2つの標的部位を同時に編集できること、(2) マウス神経芽細胞腫由来細胞においてもゲノム編集が可能なこと、(3) S. thermophilus 由来Cas9もゲノム編集に利用できることも示された。また、2012年に報告されていた短縮型sgRNAはcrRNA:tracrRNAに比べてゲノム編集効率が低いことが明らかになった。2013年7月、Zhangらは、tracrRNA tailをふくむsgRNAは、crRNA:tracrRNAや短縮型sgRNAに比べて、ヒト培養細胞における発現量や安定性が高く、効率的にゲノム編集を引き起こすことを報告した16。
Fengに聞いてみたところ、2011年1月にCas9をゲノム編集ツールとして利用するアイデアを思いつき、S. thermophilus 由来Cas9を用いて実験を開始したとのことだった。2011年のNature 論文を読み、tracrRNAはcrRNAの成熟化だけでなく、crRNA:tracrRNAとしてCas9に結合して機能するだろうと予想し、Cas9、crRNA、tracrRNAを用いて実験を進め、ゲノム編集技術の開発に成功したとのことだった。
2013年1月には、Doudna、George M. Church、J. Keith Joung、Jin-Soo KimのグループからもCRISPR-Cas9を利用したゲノム編集の成功例が相次いで報告された。急速にCRISPR-Cas9がゲノム編集に応用されたのには理由があった。1990年代からの地道な研究によって、ZFNやTALENといった人工ヌクレアーゼを利用したゲノム編集技術が確立していたからである。人工ヌクレアーゼはDNA認識モジュールとDNA切断酵素の融合タンパク質であるため、標的配列ごとにDNA認識モジュールを設計する必要があった。DNA認識モジュールの設計に比べ、sgRNAのガイド配列の変更は簡単なため、CRISPR-Cas9を用いたゲノム編集技術は瞬く間に普及した。
CRISPR-Cas9の構造解析
2012年12月、Fengから濡木理教授のもとに「CRISPR-Cas9の構造解析」に関する共同研究の誘いのメールが届いた。Fengがスタンフォード大学のKarl Deisseroth研究室の大学院生だった当時、チャネルロドプシン (光遺伝学ツール) の構造解析に関して濡木研究室と共同研究を行った経緯があった19。このような縁がCRISPR-Cas9の共同研究につながった。筆者はタンパク質-核酸複合体に興味があり、また、挑戦的な研究テーマを模索していたところだったので、すぐに構造解析を開始した。試行錯誤の末、2014年2月、Cas9-sgRNA-標的DNA複合体の立体構造を世界にさきがけて報告し、CRISPR-Cas9の作動機構の理解に貢献することができた20 (日本語解説:http://first.lifesciencedb.jp/archives/8416)。結晶構造は生化学的解析の結果とよく一致しており、(1) sgRNAのガイド配列 (スペーサー領域) と標的DNAはRNA-DNAヘテロ2本鎖を形成し、Cas9によって配列非特異的に認識されること、(2) crRNAのリピート領域はtracrRNAのアンチリピート領域と対合し、Cas9によって認識されること、(3) tracrRNA tailは2つのステムループを形成しCas9によって認識されること、(4) 2つのヌクレアーゼドメイン (RuvCとHNH) は標的DNAの切断に適した位置に存在することなど多くの知見が得られた (図2)。さらに、この研究を基盤として、異なる細菌に由来するCas9の構造解析やCas9改変体の開発といったさまざまな研究を展開することができた。
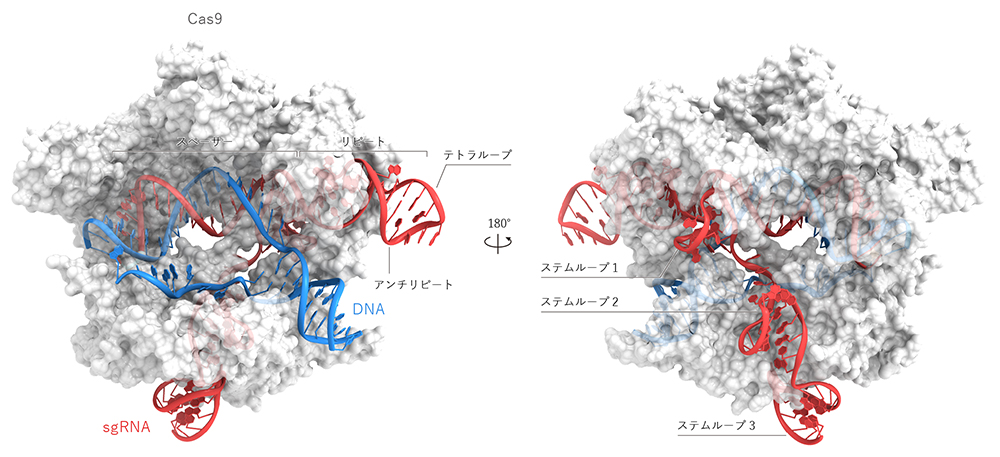
図2 Cas9-sgRNA-標的DNA複合体の結晶構造
PDB:4OO8(Cas9とsgRNA)とPDB:5F9R(DNA)を用いて作製した。
これらの研究業績が評価され、第16回 (令和元年度) 日本学術振興会賞・日本学士院学術奨励賞をいただいた。チャンスを与えてくれ、常に研究を後押ししてくれた濡木理先生、石谷隆一郎先生をはじめとする先生方に心より感謝したい。CRISPRとの出会いは人生の転機となった。理学部3号館の実験室でCas9タンパク質を精製したり、in vitro転写した何種類ものガイドRNAを巨大な変性ポリアクリルアミドゲルを用いて電気泳動したり、結晶化スクリーニングを行っていた日々がつい最近のことのように思い出される。
追記:2020年8月から東京大学先端科学技術研究センターにおいて研究室を主宰することになりました。CRISPR-Casなどのタンパク質-核酸複合体の機能解析や構造解析に興味のある学生、ポスドクを募集しています。あたらしい環境でいっしょに研究してみたい方は「nisimasu[at]g.ecc.u-tokyo.ac.jp」にご連絡ください (図3)!

図3 西増研究室 (2020年11月25日撮影)
参考文献
1. Pausch, P., Al-Shayeb, B., Bisom-Rapp, E., Tsuchida, C. A., Li, Z., Cress, B. F., Knott, G. J., Jacobsen, S. E., Banfield, J. F. & Doudna, J. A. CRISPR-CasΦ from huge phages is a hypercompact genome editor. Science 369, 333-337 (2020).
2. Ishino, Y., Shinagawa, H., Makino, K., Amemura, M. & Nakata, A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol 169, 5429-5433 (1987).
3. Makarova, K. S., Wolf, Y. I., Iranzo, J., Shmakov, S. A., Alkhnbashi, O. S., Brouns, S. J. J., Charpentier, E., Cheng, D., Haft, D. H., Horvath, P., Moineau, S., Mojica, F. J. M., Scott, D., Shah, S. A., Siksnys, V., Terns, M. P., Venclovas, C., White, M. F., Yakunin, A. F., Yan, W., Zhang, F., Garrett, R. A., Backofen, R., van der Oost, J., Barrangou, R. & Koonin, E. V. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol 18, 67-83 (2020).
4. Jansen, R., Embden, J. D., Gaastra, W. & Schouls, L. M. Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol 43, 1565-1575 (2002).
5. Mojica, F. J., Diez-Villasenor, C., Garcia-Martinez, J. & Soria, E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol 60, 174-182 (2005).
6. Barrangou, R., Fremaux, C., Deveau, H., Richards, M., Boyaval, P., Moineau, S., Romero, D. A. & Horvath, P. CRISPR provides acquired resistance against viruses in prokaryotes. Science 315, 1709-1712 (2007).
7. Brouns, S. J., Jore, M. M., Lundgren, M., Westra, E. R., Slijkhuis, R. J., Snijders, A. P., Dickman, M. J., Makarova, K. S., Koonin, E. V. & van der Oost, J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960-964 (2008).
8. Westra, E. R., van Erp, P. B., Kunne, T., Wong, S. P., Staals, R. H., Seegers, C. L., Bollen, S., Jore, M. M., Semenova, E., Severinov, K., de Vos, W. M., Dame, R. T., de Vries, R., Brouns, S. J. & van der Oost, J. CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3. Mol Cell 46, 595-605 (2012).
9. Jackson, R. N., Golden, S. M., van Erp, P. B., Carter, J., Westra, E. R., Brouns, S. J., van der Oost, J., Terwilliger, T. C., Read, R. J. & Wiedenheft, B. Crystal structure of the CRISPR RNA-guided surveillance complex from Escherichia coli. Science 345, 1473-1479 (2014).
10. Mulepati, S., Heroux, A. & Bailey, S. Crystal structure of a CRISPR RNA-guided surveillance complex bound to a ssDNA target. Science 345, 1479-1484 (2014).
11. Zhao, H., Sheng, G., Wang, J., Wang, M., Bunkoczi, G., Gong, W., Wei, Z. & Wang, Y. Crystal structure of the RNA-guided immune surveillance Cascade complex in Escherichia coli. Nature 515, 147-150 (2014).
12. Garneau, J. E., Dupuis, M. E., Villion, M., Romero, D. A., Barrangou, R., Boyaval, P., Fremaux, C., Horvath, P., Magadan, A. H. & Moineau, S. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature 468, 67-71 (2010).
13. Deltcheva, E., Chylinski, K., Sharma, C. M., Gonzales, K., Chao, Y., Pirzada, Z. A., Eckert, M. R., Vogel, J. & Charpentier, E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471, 602-607 (2011).
14. Lander, E. S. The Heroes of CRISPR. Cell 164, 18-28 (2016).
15. Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A. & Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012).
16. Hsu, P. D., Scott, D. A., Weinstein, J. A., Ran, F. A., Konermann, S., Agarwala, V., Li, Y., Fine, E. J., Wu, X., Shalem, O., Cradick, T. J., Marraffini, L. A., Bao, G. & Zhang, F. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol 31, 827-832 (2013).
17. Gasiunas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A 109, 2579-2586 (2012).
18. Cong, L., Ran, F. A., Cox, D., Lin, S., Barretto, R., Habib, N., Hsu, P. D., Wu, X., Jiang, W., Marraffini, L. A. & Zhang, F. Multiplex genome engineering using CRISPR/Cas systems. Science 339, 819-823 (2013).
19. Kato, H. E., Zhang, F., Yizhar, O., Ramakrishnan, C., Nishizawa, T., Hirata, K., Ito, J., Aita, Y., Tsukazaki, T., Hayashi, S., Hegemann, P., Maturana, A. D., Ishitani, R., Deisseroth, K. & Nureki, O. Crystal structure of the channelrhodopsin light-gated cation channel. Nature 482, 369-374 (2012).
20. Nishimasu, H., Ran, F. A., Hsu, P. D., Konermann, S., Shehata, S. I., Dohmae, N., Ishitani, R., Zhang, F. & Nureki, O. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell 156, 935-949 (2014).



