2020年のノーベル化学賞は欧米の2人の女性科学者に贈られた。その理由はRNA誘導型のヌクレアーゼを利用して、実用的なゲノム編集技術を開発したことである。本稿では、この技術に使われているCRISPRを発見した筆者が、30数年前の発見当時を顧みながら、現在までの遺伝子工学技術発展の実感を綴りたい。
はじめに
ドイツのマックスプランク研究所に所属するEmmanuelle Charpentierと、アメリカのカリフォルニア大学バークレイ校のJennifer Doudnaは、共同で実用的なゲノム編集技術を開発し、その貢献が認められたことが今回のノーベル化学賞受賞の理由である。ゲノム編集というのは、生きた細胞の遺伝情報が載ったゲノムDNAを操作して、自在に改変できる技術であり、その生物の性質を変えてしまうことができるものである。今後の人類の生活を変えてしまうような生命科学の新技術の開発の意義は極めて高いと言える。機能のわからない遺伝子の働きを解明するためにたいへん有用であり、ゲノム配列を解読した後のポストゲノム時代の生命科学にとって、ゲノム編集は間違いなく主役となる手法を提供する。本稿では、RNA誘導型ヌクレアーゼを利用した実用的なゲノム編集技術に繋がったCRISPRの発見の頃を振り返ってその感慨に浸ってみたい。
CRISPRの発見
CharpentierとDoudnaが開発したゲノム編集技術は、バクテリアやアーキアの獲得免疫システムを利用したもので、CRISPRから転写されたcrRNA (クリスパーRNA) を使ってCas9ヌクレアーゼをゲノム中の標的DNAの場所に運び、そこで狙った遺伝子を切断する技術である。CRISPRというのは、筆者が30数年前に行っていた研究から発見した独特の繰り返しDNA配列のことである (図1)。私は大阪大学微生物病研究所の中田篤男研究室 (当時の化学療法部門) で (写真1)、当時利用されはじめたばかりの、遺伝子操作技術を使って、大腸菌のある遺伝子の解析を行っていた (写真2)。そのときにそれまで見たことがない、規則正しいDNAの繰り返し配列を見つけた。それは、DNA配列としての二回対象性を含んだ29塩基が一定の間隔で5回も繰り返すというもので、この特徴は当時まだDNAの塩基配列データ量が乏しかったものの、まったく前例のないものであった1)。
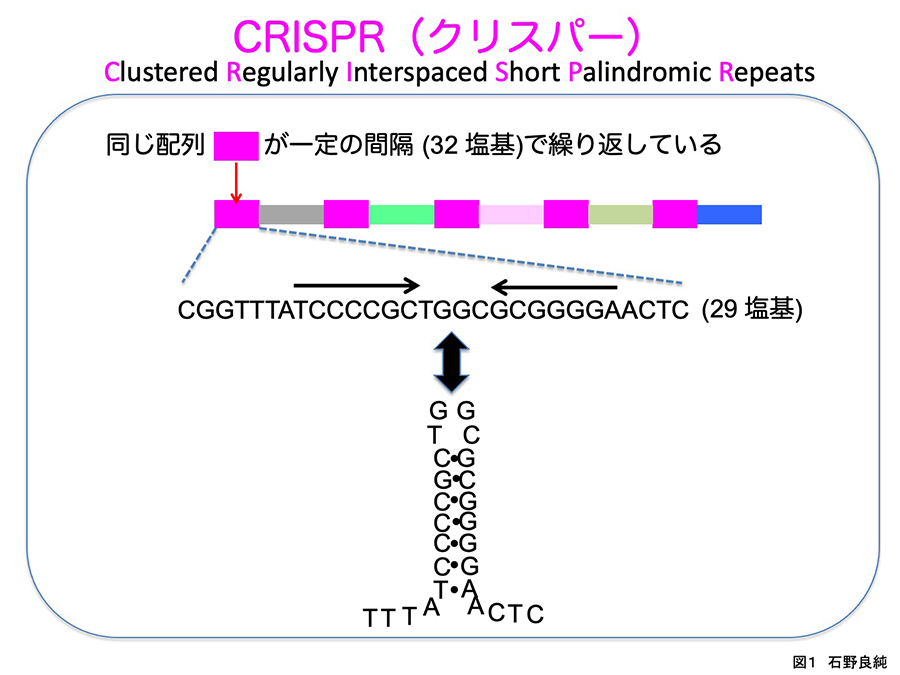
図1 CRISPR配列の特徴
筆者が発見したCRISPRは、二回対称性を含む29ヌクレオチドが一単位として、32ヌクレオチド長を挟んで等間隔に5回繰り返すものであった。

写真1 当時の大阪大学微生物病研究所
矢印の位置が化学療法部門

写真2 当時の研究室での様子
筆者と同世代の分子生物学者の方は経験されたと思うが、当時の塩基配列解読方法では、解読したいDNAを制限酵素で細かく切り刻んで、それぞれの断片をM13ファージベクターにクローニングしてから、解読操作をする必要があった。さらに、2回対称性配列はDNA鎖が自分自身で塩基対を形成してステム構造を形成してしまうために非特異的なシグナルに悩まされ、正確な解読が難しかった。この問題を克服するためには多大の苦労を要した。この繰り返し配列を含むDNAを、上の鎖と下の鎖を独立に解読して、最後に100%A-T、G-Cがマッチした時には大きな達成感があったことを記憶している。配列解読操作の過程で、異なるクローンから何度も同じ配列が出てくることは気づいていたが、最終的に解読し終えた後で改めて配列を眺めてみて、あまりに綺麗に繰り返し配列が等間隔に並んでいることに見とれてしまった。これは、細胞が生きるための何かの機能を有するに違いないと思ったが、その生物学的な意味を具体的に予想することは当時の情報からは不可能であった。
CRISPRの広がり
このような特徴をもつDNA配列はその後、大腸菌のみならず他のバクテリアやアーキアからも発見されたことで、固有の生物のものではなく、もっと広い生物学的機能を担うものと考えられるようになっていった。塩基配列解析法の進歩によって、一種の生物がもつゲノムの全配列を解読するゲノム時代になり、1990年代後半には塩基配列データの蓄積が急速に進んだ。筆者はその頃、まだ分子生物学が進んでいなかったアーキアに夢中になっていた。アーキアとして初の全ゲノム配列が報告されたのは1996年のことであるが、Craig VenterのTIGR (The Institute for Genomic Research) が、メタン産生アーキアの一種であるMethanococcus jannaschii の全ゲノム配列を解読した報告がScience誌に掲載された2)。この論文ではCRISPRが18コピーも存在することが記載されているが、ゲノム上の位置をもとにゲノムDNAの複製後の分配に関係しているのではないかという記載とともに数行触れられているに過ぎず、CRISPRという名前はまだついていなかったし、注目はされなかった。私が興味をもっていたのはアーキアのDNAポリメラーゼであり、アーキアが真核生物のDNA複製酵素であるPolα、Polδ、PolεというファミリーB型のDNAポリメラーゼで複製しているのではないかということだった。M. jannaschiiのゲノム中には、ファミリーBのDNAポリメラーゼをコードする遺伝子が一つだけ見つかり、それ以外にDNAポリメラーゼ遺伝子だと推定される配列は全く見つからなかった。アーキアがファミリーBのDNAポリメラーゼ一つだけで生きていることが驚きであることが、Nature誌、Science誌をはじめとして、関連ジャーナルでトピックスとして取り上げられた。筆者はその時、既存のDNAポリメラーゼとは全く異なる新規のDNAポリメラーゼを別のアーキアから見つけていて、報告しようとしていたが、投稿してもEditorに信じてもらえず、却下が続いて苦闘していた時だった。配列の相同性が全くないからDNAポリメラーゼだとは信じられないと露骨に言われたジャーナルもあった。M. jannaschii のゲノム中には、その新規DNAポリメラーゼと相同な配列をコードする遺伝子があることを確認し (2つのタンパク質から構成されるので遺伝子は2つ)、M. jannaschii のDNAポリメラーゼは一つではないと確信した。それは世界で我々しか知らないことだったので、たいへん夢中になってこの研究に没頭した3, 4)。CRISPRのことはすぐに忘れてしまった。
CRISPRの機能解明
スペインのAlicante大学で博士を取得するために、超好塩性アーキアが高い塩濃度の環境に適応するための遺伝子発現制御機構を研究していたFrancisco Mojicaが1993年に発表した論文の中で、アーキアにもCRISPRが存在することを見つけて記載しているが、やはり生物学的な役割はわからないと述べている5)。そしてゲノム解析時代に入った1990年代後半から急激にDNA配列データの蓄積が増した。21世紀に入り、この繰り返し配列にCRISPR (Clustered Regularly Interspaced Short Palindromic Repeat) という名称と共に、この配列の近傍に必ず存在する遺伝子群があることも発見されて、cas (CRISPR-associated) 遺伝子という名称が提唱された6)。CRISPRとcasは共同して何かの機能を担うのだろうと想像されたが、まだその役割についてはわからなかった。
博士を取得した後のMojicaは、研究費が乏しく実験が出来ずに、コンピューターを使って、データベース中のCRISPR配列と相同な配列の検察を続けていたと自らの総説論文の中で記載している7)。そして遂に、繰り返し配列の間を隔てるスペーサーの部分に、外からその細胞を攻撃して侵入してくるバクテリオファージやプラスミドの遺伝子断片が含まれている例を発見したことが、CRISPRとcas 遺伝子の機能解明の手がかりになった (図2)8)。この発見により、CRISPR-cas は、外来DNAから細胞をも衛る生体防御の役目を果たすことが予想され、それは間もなく実験で証明された。チーズ、ヨーグルトなどの乳製品を製造する工業用のStreptococcus thermophilus 菌のCRISPRの中にファージのDNAが挿入されていると、そのファージに感染しなくなり、CRISPRの中からウイルスDNAを取り除くと感染することが示されて、CRISPR-cas の機能がついに明らかになった (図3)9)。その後すぐに、CRIPSRの中に取り込まれているDNAと同じ配列のDNAをCasタンパク質が切断することで、CRISPR/Casが機能を発揮することを証明したのは、筆者のアーキア研究仲間の一人であるオランダのWageningen 大学の John van der Oostであった10)。

図2 CRISPRの生理的機能解明のヒント
CRISPRがどのような働きをするのかを解明するきっかけとなったのは、DNA配列データベースの蓄積により、繰り返し単位を挟むスペーサーの部分に、外から細胞を攻撃して侵入してくるウイルス (ファージ) やプラスミドの配列が挿入されていることが発見されたことであった。
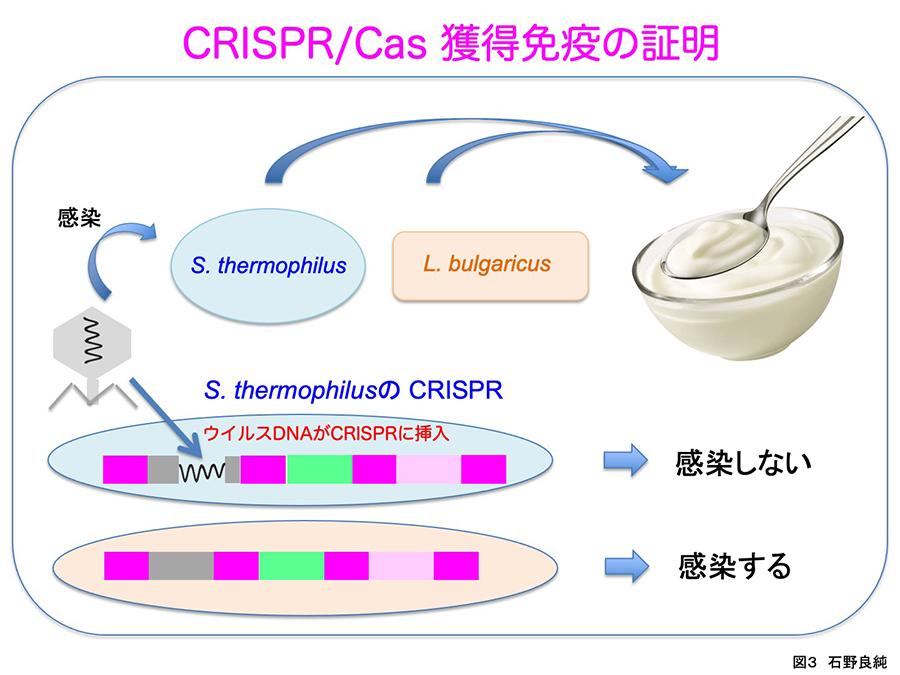
図3 CRISPR/Casによる獲得免疫機能の証明
ヨーグルトの生産菌の一つであるStreptococcus thermophilus 菌に感染するファージの遺伝子断片が、CRISPRの中に挿入されている場合は感染が起こらず、挿入されていないと感染して死滅することが実験で示された。
CRISPRのゲノム編集への応用
CharpentierとDoudnaは、CRISPR-cas が侵入してきたウイルス、ファージ、プラスミドDNAを選択的に切断するという機構を応用して、ゲノム編集に使えるという実験結果を示した。cas にはいろいろな種類があるが、Cas9は単独のタンパク質としてDNAを切るはさみの働きをすること、そして、CRISPRの配列を人工的に操作することで、ウイルスに限らず、どんな遺伝子でも狙ったところへはさみのCas9を連れていくことができることを実験で示し、任意の遺伝子を標的としてゲノム編集が可能であると明記して論文発表した。Science誌に掲載されたこの論文が、今回のノーベル賞の受賞に繋がったと言える11)。
ゲノム編集技術のコンセプトは、ゲノムDNAの狙った位置で選択的に二本鎖切断を起こすことである。切断後は、本来細胞が有するDNA修復機能を用いて、切断点を再結合する際に、欠失や挿入を入れることができる。制限酵素のように、特定の位置を切断できるヌクレアーゼやDNAリガーゼが発見されたことで、試験管内でDNAを切り貼りする遺伝子操作技術が1970年代に実用化されたが、制限酵素には認識配列が必要であり、任意の位置で切断できない。しかし、制限酵素を利用して、任意のDNA配列を切断できるZFNやTALENという人工ヌクレアーゼが開発され、ゲノム編集に使われてきた。しかし、これらはヌクレアーゼタンパク質を、個々の標的配列ごとに人工創製しなければならない。タンパク質の創製は簡単でないことは明らかである。CRISPR/Cas9の利点は、標的配列と塩基対をつくるRNA (クリスパーRNA, crRNA) が、Cas9ヌクレアーゼをその位置にリクルートすること、すなわちCas9はRNA誘導型の二本鎖切断ヌクレアーゼである (図4)。20塩基程度の短いオリゴRNAは今や簡単に化学合成できるので、ゲノム編集の汎用性が一気に高まった。

図4 CRISPR/Cas9を使った実用的ゲノム編集技術
Cas9は二本鎖DNAを切断するヌクレアーゼであるが、複合体を形成するクリスパーRNA (crRNA)が、自らの配列と相同な配列を有するDNA塩基対を形成することで、Cas9をその部位に選択的にリクルートし、その位置で切断する。
遺伝子工学技術の発展を顧みて
20世紀後半からの生命化学の急激な発展は、遺伝子工学技術の開発と進歩によるものである。DNAを試験管内で切り貼りして操作し、生きた細胞に導入する技術が開発され、その操作はPCRの誕生によって一気に簡便化した。ゲノム編集がCRISPR/Cas9の利用によって簡便化したことと類似している。
筆者は修士課程で制限酵素の研究をし12, 13)、博士論文ではDNAリガーゼ遺伝子のクローニングと多量発現系構築の研究を行った14)。また、PCR技術を日本に導入して普及に努めたバイオテク企業に、ちょうどその時期在籍し、DNAポリメラーゼを始めとするPCR関連技術開発研究に従事した15, 16)。そして、この実用的ゲノム編集技術の原点になったCRISPRの発見者ということで、この30数年の遺伝子工学技術の発展のキーテクノロジーを間近で体感してきた。初期の遺伝子操作は手慣れるための十分な経験が必要であったが、PCRはそれを塗り替えた。PCR技術そのものは極めて単純で、遺伝子やDNAポリメラーゼを扱ってきた研究者なら、何故こんな簡単なことに気がつかなかったのかと思われるだろう。筆者自身も、試験管内で鋳型、プライマーとDNAポリメラーゼを用いて、プライマーを伸長させる反応でDNA合成を行っていた経験があるので、逆向きプライマーを設定することを何故思いつかなかったのかと思い出される。CRISPR/Cas9を使うゲノム編集も、今や、高校生が生物学の実習として体験できるほど簡単な試薬セットが販売されている。技術は簡単であるほど実用的である。
CRISPR/Cas9を使った実用的なゲノム編集技術開発の成功は見事の一言に尽きる。CRISPR/Cas9が司る反応は、核酸酵素の研究者である筆者にはたいへん面白い対象であり、この原点のCRISPR発見者として、筆者が紹介されることはたいへん光栄なことである。しかし、夢中で別の研究に携わってきた筆者にとって、この反応をゲノム編集に利用するところまでまったく考えられなかった。Charpentier、Doudnaとは独立に、ほぼ同時にCRISPR/Cas9を使って、任意の標的DNAを切断できることを示したリトアニアVilnuis大学のVirginijus Siksnys (CRISPR/Cas9のゲノム編集への応用でCharpentier、Doudnaとともに、2018年のカブリ賞を受賞) は17)、筆者と同世代の核酸酵素研究者で、昔から制限酵素の研究などで活発であった。彼がCRISPR/Cas9の実用的応用に成功したことは、同じ分野の研究者としてほんとに祝福したいと思う。
ゲノム編集技術は現在、生命科学分野で急速に普及しており、基礎細胞生物学者の間では日常的に使われている。CRISPR/Casを使ったゲノム編集は、従来の遺伝子操作法と比べてより正確、より安全で、効率の良い手法である。しかしながら、技術には100%ということはなく、目的の編集だけがなされていることをできるかぎり確認する必要があるし、遺伝子組換え食品に対する一般的な嫌悪感や、デザイナーベビーの誕生が可能になる倫理的問題など、社会的に受け入れ難いマイナス点もあり、初期の遺伝子組換え実験でやられてきたように、きっちりとした規則を作り、その指針に基づいて実施するという規制は必須である。その上で、この遺伝子操作技術によって人類にもたらされる恩恵を歓迎すべきであると思う。
私が30数年前にCRISPRを発見した際に、何をするために存在するのかまったく予測がつかなかったものが、年月を重ね、多くの研究者の努力によってその獲得免疫機能が解明され、その分子メカニズムを利用してゲノム編集技術が開発されたことを振り返ると、たいへんに感慨深いものがある18)。私自身がCRISPRの機能解明を行うことができなかったのは残念であるが、このような人類に役立つすばらしい技術が誕生したことを心より歓迎したい。
おわりに
本稿で述べてきた生命科学の重要な基本技術はすべて、バクテリアやアーキアという原核微生物の基礎研究から生まれた。現在の分子生物学は、直接にヒトを対象とした研究を重視して推進されているが、原核微生物の分子生物学から人類は遺伝子操作技術を手にし、実用的なゲノム編集技術はその究極にも近い発展と言える。人類はこの技術によって多大の恩恵を受けるであろう。しかし、バクテリア、アーキアの基礎分子生物学研究を行ってきた筆者にとっては、まだまだ宝石が埋まっていることを感じている。今後の研究によって、また新しい有用技術が誕生することを期待したい。
引用文献
- Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. (1987) Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J. Bacteriol. 169, 5429-5433.
- Bult CJ. White O, Olsen GJ, Zhou L, Fleischmann RD, et al. (1996) Complete genome sequence of the methanogenic archaeon, Methanococcus jannaschii. Science 273,1058–1073.
- Ishino Y, Komori K, Cann I, Koga Y. (1998) A novel DNA polymerase family found in Archaea. J. Bacteriol. 180, 2232-2236.
- Cann IKO, Komori K, Toh H, Kanai S, Ishino Y. (1998) A heterodimeric DNA polymerase: evidence that members of the euryarchaeota possess a novel DNA polymerase. Proc. Natl. Acad. Sci. USA. 95, 14250-14255.
- Mojica MJ, Juez G, Rodríguez-Valera F.(1993) Transcription at different salinities of Haloferax mediterranei sequences adjacent to partially modified PstI sites. Mol. Microbiol. 9, 613–621.
- Mojica MJ, Rodríguez-Valera F. (2016) The discovery of CRISPR in archaea and bacteria. FEBS J. 283, 3162-3169.
- Jansen R, Embden JD, Gaastra W, Schouls LW. (2002) Identification of genes that are associated with DNA repeats in prokaryotes. Mol. Microbiol. 43, 1565–1575.
- Mojica FJM, Díez-Villaseñor C, García-Martínez J, Soria E. (2006) Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J. Mol. Evol. 60, 174–182.
- Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. (2007) CRISPR provides acquired resistance against viruses in prokaryotes. Science 315, 1709–1712.
- Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. (2008) Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960 –964.
- Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier EA. (2012) A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816–821.
- Ohtsuka E, Ishino Y, Ibaraki K, Ikehara M. (1983) Recognition by restriction endonuclease EcoRI of deoxyoctanucleotides containing modified sugar moieties. Eur. J. Biochem. 139, 447-450.
- Ishino Y, Shibahara S, Ohtsuka E, Ikehara M. (1986) Restricted joining of a decadeoxyribonucleotide for preparation of an eicosadeoxyribonucleotide duplex containing recognition sites for restriction enzymes. Nucleosides and Nucleotides 5, 471-480.
- Ishino Y, Shinagawa H, Makino K, Tsunasawa S, Sakiyama F, Nakata A. (1986) Nucleotide sequence of the lig gene and primary structure of DNA ligase of Escherichia coli. Mol. Gen. Genet. 204, 1-7.
- Uemori T, Ishino Y, Toh H, Asada K, Kato I. (1992) Organization and nucleotide sequence of the DNA polymerase gene from the archaeon Pyrococcus furiosus. Nucleic Acids Res. 21, 259-265.
- Ishino Y, Ueno T, Miyagi M, Uemori T, Imamura M, Tsunasawa S, and Kato, I. (1994) Overproduction of Thermus aquaticus DNA polymerase and its structural analysis by ion-spray mass spectrometry. J. Biochem. 116, 1019-1024.
- Gasiunas G, Barrangou R, Horvath P, Siksnys V. (2012) Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. USA. 109, E2579–2586.
- Ishino Y, Krupovic M, Forterre P. (2018) History of CRISPR-Cas from Encounter with a Mysterious Repeated Sequence to Genome Editing Technology. J. Bacteriol. 200, e00580-17.



