本プロトコルは哺乳類培養細胞におけるribosome profilingの基本プロトコルです。HEK293T細胞を想定して記述していますが、経験上同様の手法が他の培養細胞でも適用できることが多いです。
ribosome profilingに関する相談、質問、コメント等あれば岩崎(shintaro.iwasaki [at mark] riken.jp)までご一報いただければありがたいです。Citationはこちらです(https://www.ncbi.nlm.nih.gov/pubmed/28579404)。
BASIC PROCEDURE
サンプル4本の場合
1. Lysate作製
【シクロへキシミド処理】
| [x1 sample] | [In 5 mL] | [final] | |
|---|---|---|---|
| 1M Tris-HCl pH 7.5 | 12 | 100 | 20 mM |
| 5M NaCl | 18 | 150 | 150 mM |
| 1M MgCl2 | 3 | 25 | 5 mM |
| 0.1M DTT | 6 | 50 | 1 mM |
| 10% Triton X-100 | 60 | 500 | 1% |
| RNase-free water | 500.4 | 4170 | |
| total | 600 | 5 mL |
上記のバッファーを作製し、冷やしておく。
以下の試薬を使用直前に加える。
| [In 5 mL] | [final] | |
|---|---|---|
| Cycloheximide 100 mg/mL | 5 | 100 microg/mL |
1. 10 cmディッシュの細胞を冷PBS 5 mlでリンスする。
2. PBSをよく吸い捨て、Lysis bufferを400 ul入れて全体に広げる。
3. ピペッティングで細胞を剥がし、DNA Lobind チューブに移す。Lysis buffer 200 ulで洗い込む。
4. 2 U/ul Turbo DNase I 7.5 ul 加え、氷上に10 minおく。
5. 20,000 g 4℃ 10 min
6. 上清をチューブに取り、転倒混和する。
7. 濃度測定用5 ul 1本、残りは100 ulずつ分注し、液体窒素で瞬間冷凍し-80℃で保存する。
PAUSE POINT -80℃
【RNA濃度測定Qubit RNA BR Assay Kit】
1. Working Solution 200 ul ×(スタンダード用 2 本+サンプル 4本分)を用意する。Working Solution=Qubit RNA BR Reagent 1 ul : Qubit RNA BR Buffer 200 ul
2. 0.5 mlチューブにスタンダード用190 ul 、サンプル用199 ulずつ分注。スタンダード試薬#1,#2を10ul、サンプルを1ulずつ加え、ボルテックス、スピンダウン。室温で2 min 置く。
3. Qubit 2.0 Fluorometer で測定、RNA BR Assayを選択。スタンダード、サンプルを測定する。
*80% confluentのHEK cellで10 cmディッシュ1枚から200-300 ng/ulになる。(600 ul lysateの場合)
2. RNase消化〜超遠心〜リボソームの精製
・ヒートブロック 25℃準備
・超遠心機のローター冷やしておく
| [In 5 mL] | [final] | |
|---|---|---|
| Sucrose | 1.7 g | 1M |
| 1M Tris-HCl pH 7.5 | 100 | 20 mM |
| 5M NaCl | 150 | 150 mM |
| 1M MgCl2 | 25 | 5 mM |
| 0.1M DTT | 50 | 1 mM |
| RNase-free water | 3565 | |
| Total | 5 mL |
Sucroseが溶けたら氷上で冷やしておき、使用直前に以下を加える。
| [In 5 mL] | [final] | |
|---|---|---|
| Cycloheximide 100 mg/mL | 5 | 100 microg/mL |
| SUPERase In 20U/microL | 5 | 20U/mL |
【サンプル準備】
・Rnase Iは 2 U/1ug RNAで使用する。(RNA 10 ugの時20 U使用)
| [microL] | |
|---|---|
| RNA Lysate (RNA 10 microg分) | X |
| Lysis buffer (用時調製) | Y |
| RNase I (10U/microL) | 2 |
| total | 300 |
1. チューブにRNAをとり、Lysis bufferで298 ulにする
2. RNase Iを2 ulずつ入れ優しく混合、ヒートブロックで25℃ 45 min(サンプル間で反応時間に差が出ないよう正確に).
3. 氷上へ移して、すぐに SUPERase In (RNase inhibitor)を10 ulずつ加える。 (冷やすことで反応止める効果があるので先に氷上へ)
4. 超遠心チューブにRNaseI消化後のサンプル300 ulを移す。
5. Sucrose cushion buffer 900 ulをサンプルの下から2層になるようゆっくり注ぐ。
6. TLA110 ローターで、100,000 rpm 4℃ 1 h
7. ペレットを崩さないよう、上清を液面の上部から吸い捨てる.
8. ペレットにTRIzol reagent を300 ul入れて(ペレットが見えるようになる)よく溶かす。
9. DNA Lobindチューブに移す。
PAUSE POINT -80℃
【Direct-zol MicroPrep kit カラム精製】
| [microL] | |
|---|---|
| TRIzol sample | 300 |
| エタノール(等量) | 300 |
| total | 600 |
1. カラムに移して12,000 g, 1 min, 4℃. 受けチューブは1回ごとに使い捨てる。
2. PreWash buffer 400ul を加え12,000 g, 1 min, 4℃.2回行う
3. Wash Buffer 700 ul, 12,000 g, 1 min, 4℃.
4. 空遠心 12,000 g, 5 min, 4℃.
5. RNase-free waterを6 ul を加え 12,000 g, 2 min, 4℃.
6. 2x RNA Loading Bufferを6 ul加える
PAUSE POINT -80℃
3. Footprint Fragment 精製
【RNA サイズマーカー準備と泳動】
| 2レーン分 | [microL] |
|---|---|
| 10 microM NI800 (34 nt) | 1 |
| 10 microM NI801 (26 nt) | 1 |
| RNase-free water | 10 |
| 2x RNA Loading Buffer | 12 |
| total | 24 |
1. RNA サイズマーカーとサンプルをヒートブロックで95℃ 3 min → 氷上 2 min
2. WAKO SuperSep RNA 15% ゲルを用意
3. well掃除を行ってから、サンプルをアプライする。コンスタント10 mA で50 min泳動する。
4. 1 x TBE 50 ml + SYBRGold 5 ul (10,000倍希釈)でゲルを染色する。シェーカーで揺らしながら3 min
5. ブルーライトに乗せて、バンドを確認しサンプルの26bp-34bpの間を切り出す。
6. マーカーも切り出しておく。
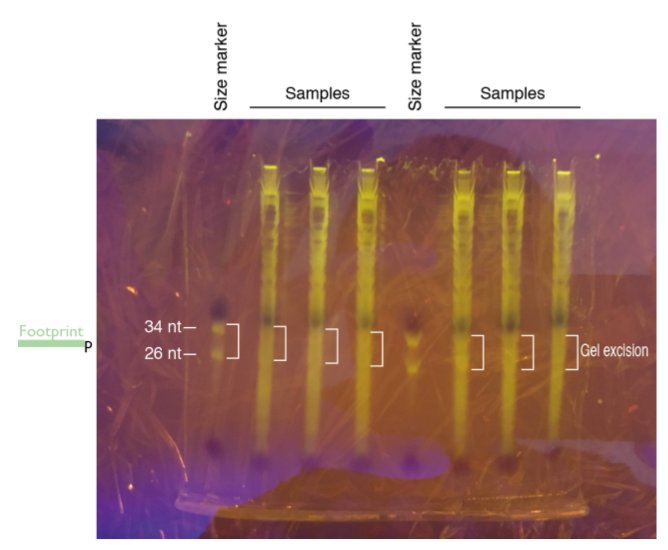
【gelからのRNA抽出】
| [microL] | [final] | |
|---|---|---|
| 3M 酢酸Na pH 5.2 | 400 | 300 mM |
| 0.5M EDTA | 8 | 1 mM |
| 10% SDS | 100 | 0.25% |
| RNase-free water | 3492 | |
| total | 4000 |
1. 1.5 mlチューブに入れたゲル片をペッスルで潰す。
2. RNA gel extraction buffer 400 ulを入れ、ペッスルについたゲルを洗い込む。
3. −80℃, 30 min or 液体窒素で凍結する。
4. 室温 2 h以上転倒混和する。
5. Spin-XカラムをDNA Lobind 1.5 mlチューブにセットし、先太チップでゲル溶液を移す。
6. 10000 g, 1 min, 4℃
7. GlycoBlue 3 ul , イソプロパノール500 ulを加えよく混ぜる。
8. 冷凍庫に1 h入れた後、20,000 g, 30 min, 4℃.
9. 上清を捨てて、70% エタノールリンス
10. ペレットに10 mM Tris pH7.5を7 ul入れ、溶かす。
PAUSE POINT -80℃
4. 20 uM Preadenylylated linker 作製
1. 以下の溶液を8連チューブに調製する。
| [microL] | |
|---|---|
| 100 microM 5′p-linker-ddC primer | 1.2 |
| 10x 5′DNA Adenylation reaction buffer | 2 |
| 1 mM ATP | 2 |
| Mth RNA Ligase | 2 |
| RNase-free water | 12.8 |
| total | 20 |
65℃ 1 h → 85℃ 5 min
2. Oligo clean & Concentrator カラム精製
| [microL] | |
|---|---|
| サンプル20 microL + RNase-free water 30 microL | 50 |
| Binding buffer(2倍量) | 100 |
| total | 150 |
| Mix | + |
| エタノール(8倍量) | 400 |
| total | 550 |
カラムに移して12,000 g, 1 min.
Wssh buffer 750 ul を加え、12,000 g, 1 min.
空遠心 12,000 g, 5 min.
RNase-free water 6 ulでElution.
PAUSE POINT -20℃
5. Dephosphorylation and Linker Ligation
【脱リン酸化】
1. Mixtureの準備 マーカー + サンプル4つの場合
| [x1 sample] | [x5.2 (premix)] | |
|---|---|---|
| RNA sample | 7 | - |
| 10x T4 PNK buffer | 1 | 5.2 |
| T4 PNK | 1 | 5.2 |
| SUPERase In | 1 | 5.2 |
| total | 10 | 15.6 |
2. サンプルは8連チューブに移し、95℃2 min → 氷上3 min
3. mixtureを3ulずつ分注し 37℃ 1 h
PAUSE POINT -80℃
【リンカーライゲーション】
・サンプルごとにそれぞれ別のリンカーをつける。メモしておく。
・マーカーにはどのリンカーをつけても良い
1. 脱リン酸化後 95℃2 min → 氷上3 min
| [x1 sample] | [x5.2 (premix)] | |
|---|---|---|
| 50% PEG-8000 | 7 | 36.4 |
| 10x T4 RNA ligase buffer | 1 | 5.2 |
| T4 Rnl2(tr)K227Q (200U/microL) | 1 | 5.2 |
| total | 9 | 46.8 |
2. サンプルに9 ulずつ分注
3. Preadenylated linker (20 uM)を1 ulずつ分注 mix
4. 22℃ 3 h → 4℃ ∞
5. Oligo clean カラム精製(前述の通りに精製)する。
6. Elution 6 ul + 2 x RNA Loading Buffer 6 ul
PAUSE POINT -80℃
【泳動準備】
① linker のみ
| [microL] | |
|---|---|
| Preadenylated linker (20 microM) | 1 |
| RNase-free water | 5 |
| 2x RNA Loading Buffer | 6 |
| total | 12 |
② Linker ligation後のマーカー
③ Linker ligation後サンプル
1. ヒートブロック 95℃ 3 min → 氷上 2 min
2. 前述の通りに泳動する。
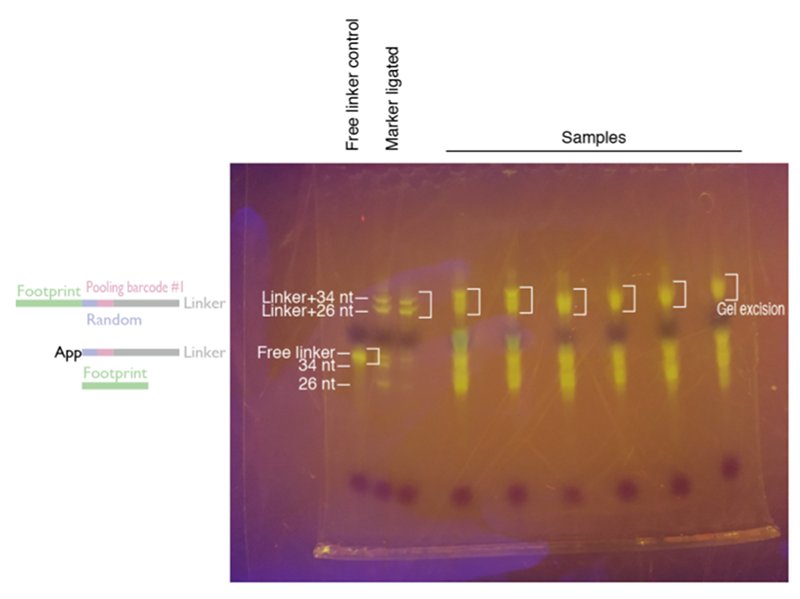
3. Linker ligation後のサンプルとマーカーを切り出す。
*切り出したサンプルはリンカーがそれぞれ違うので、以降は混ぜても良い。
4. gelからのRNA抽出(前述の通り)
〜70%リンス後ペレットを溶解時〜
次のRibo-Zero処理では4サンプルをまとめて処理する。全量が26 ulになるように、10 mM Tris pH 7.5で溶解する。
マーカーはRibo-Zero処理しないので、10 mM Tris pH7.5を10 ulで溶解する。
PAUSE POINT -80℃
6. Ribo-Zero処理
1. ビーズを225 ul チューブに移し、マグネットに立てる。
2. 上清を捨てて、RNase-free water 225 ul で2回洗浄する。
3. Resuspension solution 60 ulで懸濁する。室温においておく。
4. 1.5mlチューブに以下の溶液を作製。
| [microL] | |
|---|---|
| RNAサンプル(4サンプル分) | 26 |
| Ribo-Zero reaction buffer | 4 |
| rRNA Removal SIn-Gold | 10 |
| total | 40 |
68℃ 10 min → 室温 5 min
5. 用意しておいたビーズ65 ulを入れピペッティング、vortex 10 s。室温で5 minおいた後vortex 10 s、マグネットに立てる。
6. 上清を新しいチューブに移す。
7. Oligo clean & Concentratorカラム精製を行う。
| [microL] | |
|---|---|
| Ribo-Zero後サンプル | 100 |
| binding buffer(2倍量) | 200 |
| total | 300 |
| Mix | + |
| エタノール(8倍量) | 800 |
| total | 1100 |
(カラムには800 ulまでしか入らないので、2回に分けてカラムに通す)
前述の通りに精製し、RNase-free water 10 ulでElution。
PAUSE POINT -80℃
7. 逆転写反応
1. 8連チューブに以下のものを用意する。
① RT primerのみ (RNase-free water 10ul) or
② RT primer + linker (RNase-free water 9.5 ul + 20 uM liker 0.5 ul) or
③ マーカー(linker ligation後)10 ul or
④ サンプル(linker ligation、Ribozero後)10 ul
+
1.25 uM RT primer NI802 2 ul
total 12 ul
65℃ 5 min → 氷上 5 min
2. mixtureを①〜④ + RT primerに8ulずつ分注する
| x1 | x4.2 (premix) | |
|---|---|---|
| 5x Protoscript II buffer | 4 | 16.8 |
| 10mM dNTPs | 1 | 4.2 |
| 10x DTT | 1 | 4.2 |
| SUPERase In | 1 | 4.2 |
| Protoscript II | 1 | 4.2 |
| ①〜④ + RT primer | 12 | - |
| total | 20 | 33.6 |
3. 50℃ 30 min
4. 1 M NaOHを2.2 ulずつ加え、混ぜる。70℃ 20 min
5. Oligo clean & Concentrator カラム精製
RNase-free waterを28 ul足して50 ulにする。前述の通りに精製する。
Elution 6ul + 2x RNA Loading Buffer 6ul
PAUSE POINT -20℃
【泳動準備】
・リンカーとRT primerがアニールしたものがサンプルサイズと重なるため、ヒートブロックで100℃ 5 min → 氷上においてから泳動する。
DNA gel extraction buffer(泳動の間に作っておく)
| [microL] | [final] | |
|---|---|---|
| 5M NaCl | 300 | 300 mM |
| 1M Tris pH7.5 | 50 | 10 mM |
| 0.5M EDTA | 10 | 1 mM |
| RNase-free water | 4640 | - |
| total | 5000 | - |
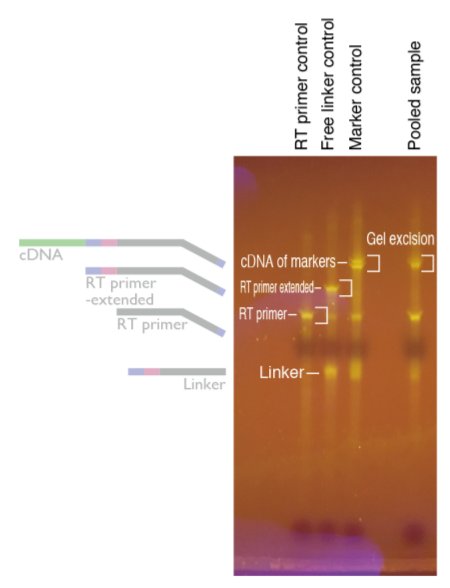
・切り出したゲルはDNA gel extraction bufferを入れDNAを抽出し、前述の通りにイソプロ沈を行い、ペレットに10 mM Tris pH7.5を12 ul入れ溶かす。
8. Circularization
1. mixtureを8連チューブに8 ulずつ分注する。
| x1 sample | x4.2 (premix) | |
|---|---|---|
| First strand cDNA | 12 | - |
| 10x CircLigase II buffer | 2 | 8.4 |
| 5M Betine | 4 | 16.8 |
| 50 mM MnCl2 | 1 | 4.2 |
| CircLigase II (100U/microL) | 1 | 4.2 |
| total | 20 | 33.6 |
2. サンプルを12 ulずつ分注する
3. 60℃ 1 h → 80℃ 10 min
PAUSE POINT -20℃
9. PCR Amplification and Barcode Addition
・サンプルはサイクル数を振って(6 ,8, 10サイクル)、非特異バンドが少ないバンドを切り出す。
・コントロール① RT primerのみ② RT primer + linker ③ マーカー(inker ligation後)は8サイクルのみでよい。
1. サンプルは反応液100 ulを作り、33 ulずつ3つのwellに分ける(6 ,8, 10サイクル)
コントロールは100 ul 分のtemplateなし反応液作成、3つのwellに分け、templateを1.7 ulずつ加える(8サイクル)。
PCR後、ライブラリーをさらにpoolする場合は、ライブラリーごとに異なるRv primer (NI822-826)を必要に応じて用いる。
(poolできるサンプル数 = linkerバーコード X PCR primerバーコード)
| サンプル | コントロール | 1 well | |
|---|---|---|---|
| 5x Phusion HF buffer | 20 | 20 | 6.7 |
| 2.5 mM dNTPs | 8 | 8 | 2.7 |
| 10 microM NI798 Fw primer | 5 | 5 | 1.7 |
| 10 microM NI799 Rv primer | 5 | 5 | 1.7 |
| Circularized cDNA template | 5 | - | 1.7 |
| H2O | 56 | 56 | 18.7 |
| Phusion polymerase (2U/microL) | 1 | 1 | 0.3 |
| total | 100 | 95 | 33 |
2. PCRをスタートさせたら、各サイクルの伸長反応が終わったところでチューブを回収していく。
1) 98℃ 30 s
2) [98℃ 10 s, 65℃ 10s, 72℃ 5 s] x6, x8, x10(になるよう2サイクルずつ回収する)
3) 72℃ 5 min
4) 4℃ ∞
PAUSE POINT -20℃
10. PCR productのゲル精製
PCR productはdenatureさせてはいけない。ゲルはSuperSep DNA, 15%を使用する。 Loading Dyeは6 x non-denaturing purple loading dyeを使用する。
【泳動準備】
・サンプル33 ul + 6 x non-denaturing purple loading dye 6.5 ul を混合し、2 wellに19 ulずつ泳動する
・RT primer, linker, Marker 33 ul + 6 x non-denaturing purple loading dye 6.5 ul を混合し、2 wellに10 ulずつ泳動する
・20 mA 1 h 20min泳動し、前述の通りにゲル染色する
・最適サイクル数を確認して、バンドを切り出す

【gelからのDNA抽出】
1. 2 well分のゲル片をペッスルで潰し、DNA gel extraction buffer 230 ul入れる。
2. -80℃ or 液体窒素で凍結後、室温2 h以上転倒混和。Spin-Xカラムでゲル片を取り除く。
3. NucleoSpin Gel and Cleanカラム精製
| [microL] | |
|---|---|
| DNA抽出液 | 230 |
| Buffer NT1(2倍量) | 460 |
| total | 690 |
4. カラムに移して、11,000 g, 1 min, 4℃.
5. Buffer NT3 700ul でWASH 11,000 g, 1 min, 4℃. x2回
6. 空遠心 11,000 g, 5 min.
7. NE buffer 17 ul 加え、室温 1 minおく
8. Elution 11,000 g, 1 min, 4℃.
11. Quality Check by MultiNA
【超高感度モードで測定】
※設定が必要なので島津に相談
・1/25 Gel Star(TEで25倍希釈したもの)
・1/5 島津マーカーDNA-1000(H2Oで5倍希釈したもの)
・1/50 100 bp DNA ラダー(TEで50倍希釈したもの)
1. 分離Buffer の準備
| [microL] | |
|---|---|
| Separation buffer (DNA-1000) | 398 |
| 1/25希釈Gel Star | 2 |
| total | 400 |
2. サンプル、ラダーの準備
| [microL] | |
|---|---|
| サンプル or 1/50希釈ラダー | 2 |
| 1/5島津マーカー | 4 |
| total | 6 |
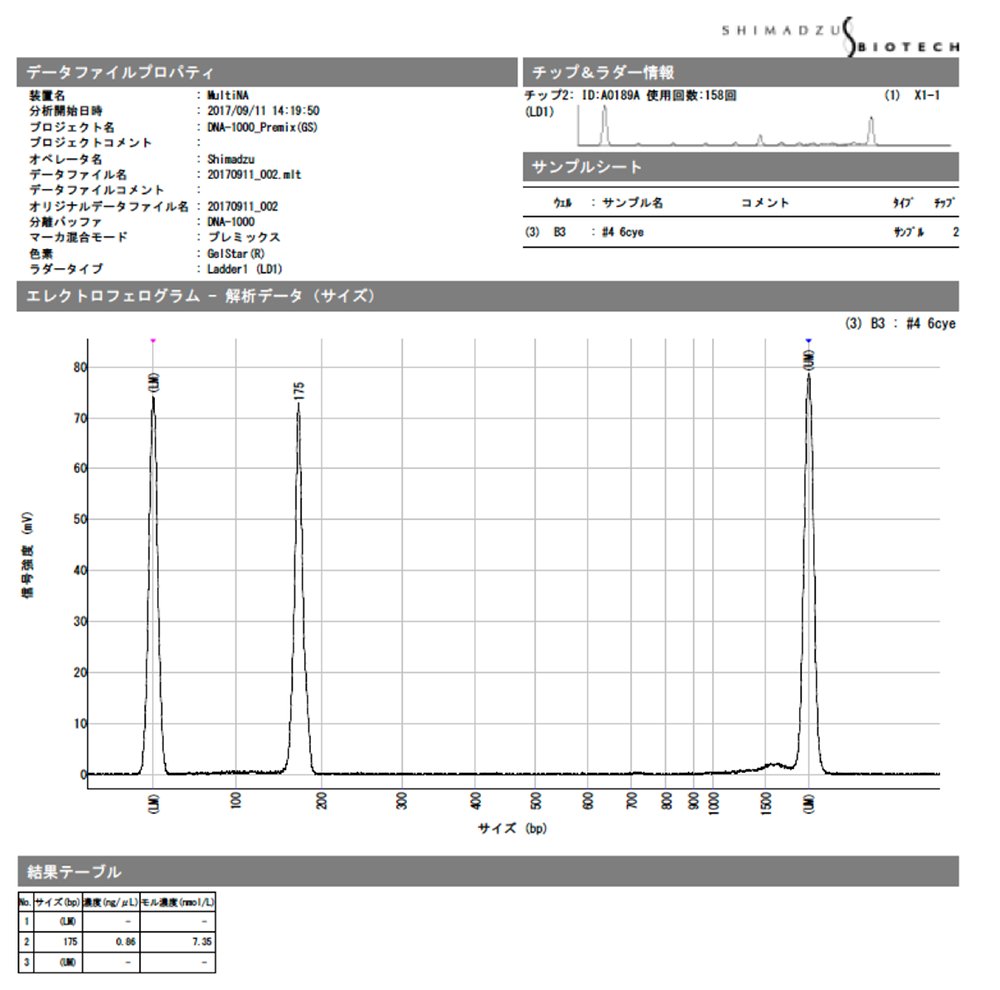
3. 機械にセットして測定
4. 濃度、mol数は結果を1/5した値になる。(超高感度測定のため)
シークエンスに必要な量は1 nM が15 ul分です。
足りない場合は最適サイクルのみを100ul分PCRし、同様にゲル精製を行ってください。
PCR Duplicateを減らすために、解析に出すサンプルはサイクル数の少ない方を優先します。サイクル数の違うサンプル同士は混ぜないでください。
Materials
l HEK293 cells (ATCC, cat. no. CRL-1573)
l DMEM (1x) + GlutaMAX-I (ThermoFisher Scientific, cat. no. 10566-016), with 10% FBS before use.
l 0.05% Trypsin-EDTA (ThermoFisher Scientific, cat. no. 25300-54)
l Cycloheximide 100 mg/ml (Sigma/Aldrich, cat. no. C4859-1ML)
l D-PBS (-)(1x) (nacalai tesque, cat. no. 14249-24)
l RNase-free water, molecular biology grade (Millipore, cat. no. H20MB1001) or (ThermoFisher Scientific cat. no. 10977-015)
l 1 M Tris-HCl pH 7.5, molecular biology grade (Wako Pure Chemical Industries, Ltd., cat. no. 318-90225)
l 5 M NaCl, molecular biology grade (nacalai tesque, cat. no. 06900-14)
l 1 M MgCl2, molecular biology grade (nacalai tesque, cat. no. 20942-34)
l Turbo DNase, 2 U/µl (ThermoFisher Scientific, cat. no. AM2238)
l Triton X-100, molecular biology grade (nacalai tesque, cat. no. 12967-32)
l Qubit RNA BR Assay kit (ThermoFisher Scientific, cat. no. Q10210)
l SUPERase·In, 20 U/µl (ThermoFisher Scientific, cat. no. AM2694)
l Sucrose, molecular biology grade (Wako Pure Chemical Industries, Ltd., cat. no. 198-13525)
l 3 M NaOAc pH 5.2, molecular biology grade (nacalai tesque, cat. no. 06893-24)
l RNase I, 10 U/µl (Epicentre, cat. no. N6901K)
l 13 x 56 mm polycarbonate ultracentrifuge tube (Beckman Coulter, cat. no. 362305)
l Direct-zol RNA MicroPrep (Zymo Research, cat. no. R2062)
l TRIzol (ThermoFisher Scientific, cat. no. 15596018) or other Direct-zol compatible reagent
l Ethanol, molecular biology grade (Wako Pure Chemical Industries, Ltd., cat. no. 054-07225)
l Isopropanol, molecular biology grade (Wako Pure Chemical Industries, Ltd., cat. no. 168-21675)
l GlycoBlue, 15 mg/ml (ThermoFisher Scientific, cat. no. AM9515)
l 0.5 M EDTA, molecular biology grade (Wako Pure Chemical Industries, Ltd., cat. no. 311-90075)
l 2× RNA Loading Buffer without Ethidium Bromide (Wako Pure Chemical Industries, Ltd. cat. no. 182-02571)
l SuperSepRNA, 15%, 17well (Wako Pure Chemical Industries, Ltd. cat. no. 194-15881)
l Upper size marker oligoribonucleotide NI800 (34 nt),
5´-AUGUACACUAGGGAUAACAGGGUAAUCAACGCGA-(Phos).
l Lower size marker oligoribonucleotide NI801 (26 nt),
5´-AUGUUAGGGAUAACAGGGUAAUGCGA-(Phos).
l 10,000x SYBR Gold (ThermoFisher Scientific, cat. no. S11494)
l UltraPure 10% SDS (ThermoFisher Scientific, cat. no. 15553-027)
l T4 polynucleotide kinase (New England Biolabs, cat. no. M0201S). Supplied with 10x T4 polynucleotide kinase buffer.
l T4 RNA Ligase 2, truncated K227Q (New England Biolabs, cat. no. M0351S). Supplied with PEG 8000 50% w/v and 10x T4 RNA ligase buffer
l Preadenylated linkers at 20 µM
l Oligo Clean & Concentrator (Zymo Research, cat. no. D4060)
l 10 mM dNTP mix (New England Biolabs, cat. no. N0447L)
l ProtoScript II (New England Biolabs, cat. no. M0368L). Supplied with 5x first-strand buffer and 0.1 M DTT
l Reverse transcription primer, NI-802,
5´-(Phos)NNAGATCGGAAGAGCGTCGTGTAGGGAAAGAG(iSp18)GTGACTGGAGTTCAGACGTGTGCTC.
l 1M Sodium hydroxide (nacalai tesque, cat. no. 37421-05)
l CircLigaseII ssDNA ligase (Epicentre, cat. no. CL9025K). Supplied with 10x CircLigaseII buffer, 5 M Betaine, and 50 mM MnCl2.
l Forward library PCR primer, NI-798,
5´- AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTC
l Indexed reverse library PCR primers,
NI-799,
5´-CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTG
l Phusion polymerase (New England Biolabs, cat. no. M0530S). Supplied with 5x HF buffer.
l Gel Loading Dye, Purple (6×) (New England Biolabs, cat. no. B7024S)
l SuperSep DNA, 15%, 17 well (Wako Pure Chemical Industries, Ltd., 190-15481)
l DNA-1000 kit (SHIMAZU BIOTECH)
l GelStar Nucleic Acid Gel Stain 10,000× (LONZA, cat. no. 50535)
l 100 bp DNA Ladder (TAKARA BIO, cat. no. 3407A)
l NucleoSpin Gel and PCR Clean-up (TAKARA, cat. no 740609.250)
Equipment
l DNA LoBind Tube 1.5 ml (eppendorf, cat. no. 022431021)
l 8-strip PCR tube with lid (BIO-BIK, cat. no. 3247-00)
l Nunc 50mL Conical Sterile Polypropylene Centrifuge Tubes (ThermoFisher Scientific, cat. no. 339652)
l Eppendorf Tubes 5.0 ml (eppendorf, cat. no. 0030122313)
l Low retention filter tips (greiner bio-one, cat. nos. 771265, 773265, 738265, and 750265)
l Short 10 µl filter tips (watson, cat. no. 1252-207CS)
l Wide Bore 200 µl filter tips (Axygen, cat. no. TF-205-WB-R-S)
l Gel loading 20 µl filter tips (ThermoFisher Scientific, cat. no. 2155P)
l Refrigerated microcentrifuge (TOMY, cat. no. MX-307)
l Qubit 2.0 Fluorometer (ThermoFisher Scientific)
l Optima MAX-TL Ultracentrifuge (Beckman, cat. no. A95761)
l TLA 110 rotor (Beckman, cat. no. 366735)
l Dry block heater (Major science, cat. no. MC-0203)
l EasySeparator (Wako Pure Chemical Industries, Ltd. cat. no. 058-07681)
l Electrophoresis power supply (Amercham Biosciences, cat. no. EPS301)
l Blue light illuminator and orage filter cover (NA). A standard UV transilluminator can be used instead.
l Razors (Feather, cat. no. FAS-10) or (Feather, cat. no. No 11 stainless steel)
l Spin-X centrifuge tube filter 0.22 µM (costar, cat. no. 8160)
l Thermal cycler (Applied Biosystems, cat. no. 2720)
l DynaMag-2 separation rack (ThermoFisher Scientific, cat. no. 12321D)
l MixMate (eppendorf)
l MultiNA (SHIMAZU BIOTECH)
l Disposable homogenizer pestle R-1.5 (ASONE, cat. no. 1-2955-01)
Custom Linker Barcodes
|
Index |
Primer |
Barcode |
Oligo sequence |
|
1 |
NI-810 |
ATCGT |
5´-/5Phos/NNNNNATCGTAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
2 |
NI-811 |
AGCTA |
5´-/5Phos/NNNNNAGCTAAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
3 |
NI-812 |
CGTAA |
5´-/5Phos/NNNNNCGTAAAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
4 |
NI-813 |
CTAGA |
5´-/5Phos/NNNNNCTAGAAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
5 |
NI-814 |
GATCA |
5´-/5Phos/NNNNNGATCAAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
6 |
NI-815 |
GCATA |
5´-/5Phos/NNNNNGCATAAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
7 |
NI-816 |
TAGAC |
5´-/5Phos/NNNNNTAGACAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
|
8 |
NI-817 |
TCTAG |
5´-/5Phos/NNNNNTCTAGAGATCGGAAGAGCACACGTCTGAA/3ddC/ |
PCR Indexing Primers
|
Index |
No. |
Oligo |
Oligo sequence |
|
ATCACG |
1 |
NI-799 |
5´-CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTG |
|
CGATGT |
2 |
NI-822 |
5´-CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGTG |
|
TTAGGC |
3 |
NI-823 |
5´-CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTG |
|
TGACCA |
4 |
NI-824 |
5´-CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTG |
|
ACAGTG |
5 |
NI-825 |
5´-CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTG |
|
GCCAAT |
6 |
NI-826 |
5´-CAAGCAGAAGACGGCATACGAGATATTGGCGTGACTGGAGTTCAGACGTGTG |







